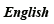سپهری, ساقی ، باخرد, زهره ، استانی, فرشته (1401) شناسایی مهارکننده های آنزیم آلفا-گلوکوزیداز بر پایه اسکلت های هیبریدی تریازول-کومارین با استفاده از تکنیک های محاسباتی طراحی دارو. دکتری حرفه ای (پایان نامه) دانشگاه علوم پزشکی اردبیل.
متن کامل
|
متنی
- گزارش نهایی طرح تحقیقاتی/ پایان نامه
8MB | |
|
متنی
- گزارش نهایی طرح تحقیقاتی/ پایان نامه
8MB | |
|
متنی
- گزارش نهایی طرح تحقیقاتی/ پایان نامه
محدود به فقط پرسنل سامانه 6MB | ||
|
متنی
- گزارش نهایی طرح تحقیقاتی/ پایان نامه
محدود به فقط پرسنل سامانه 3MB |
آدرس اینترنتی رسمی : https://lib.arums.ac.ir/cgi-bin/koha/opac-ISBDdeta...
خلاصه فارسی
مقدمه و هدف: دیابت شیرین یک اختلال متابولیسمی و چندعاملی است. آنزیم آلفا-گلوکوزیداز در اپی تلیوم روده کوچک مسئولیت هضم کربوهیدرات ها را بر عهده دارد. مهارکردن این آنزیم با جلوگیری از هضم کربوهیدراتها میتواند هایپرگلایسمی پس از مصرف غذا را کاهش دهد. طراحی و گزارش ترکیبات جدید دارای هسته کومارین و تریازول به کمک غربالگری مجازی، روشی بهینه، ساده و کمهزینه است و میتواند در مهارکردن آنزیم آلفا-گلوکوزیداز مفید واقع شود. مواد و روشها: ابتدا ساختار کریستالوگرافی آنزیم آلفا-گلوکوزیداز با کد شناساگر 3A4A از بانک داده پروتئین (PDB) دریافت گردید. سپس، کتابخانهای از ۱۹۲۲ ترکیب شیمیایی که ازنظر ساختاری مشابه با اسکلت های اصلی تریازول و کومارین بودند، از پایگاه دادهای PubChem تشکیل شد. فیلترهای مختلفی توسط نرمافزار PyRx0.8 و سرورهای Molinspiration، SwissADME و admetSAR بر روی ترکیبات اعمال گردید. درنهایت، انرژی اتصال و برهمکنش ترکیبات توسط نرم¬افزار Autodock4.2 بررسی شد. یافتهها: از میان ترکیبات موجود در کتابخانه، هشت ترکیب با موفقیت از فیلترهای مذکور عبور کردند. سپس، بر روی این ترکیبات داکینگ مولکولی انجام شد. از میان هشت ترکیب داک شده، چهارترکیب با کدهای شناسایی CID_132277006، CID_1534998، CID_110745226 و CID_122233777 به ترتیب با انرژی اتصال 9/19-، 8/89-، 8/71- و 8/49- کیلوکالری بر مول بهعنوان بهترین ترکیبات معرفی شدند. بررسی نتایج داکینگ مولکولی نشان داد که اسیدآمینههای Arg442، Gln279، Ser241، His351 ، Lys156، Asp352، Glu277، Arg315، Asp242،Phe303 ، Ser157، Asn415، Glu411، Phe314، Tyr158،Ser240 ،Asp215 و Val216 اهمیت بالاتری در تشکیل کمپلکس ترکیبات با جایگاه فعال آنزیم آلفا-گلوکوزیداز دارند. بحث و نتیجهگیری: به نظر میرسد پیوند هیدروفوب و در درجه بعدی پیوند هیدروژنی و پس ازآن پیوند کاتیون-π، جهت ایجاد برهمکنش ترکیبات با جایگاه فعال آنزیم آلفا-گلوکوزیداز مهم هستند.
عنوان انگليسی
Identification of alpha-glucosidase enzyme inhibitors based on triazole-coumarin hybrid scaffolds using computational drug design techniques
خلاصه انگلیسی
Background and Purpose: Diabetes mellitus is a multifactorial and metabolic disorder. The alpha-glucosidase enzyme in the epithelium of the small intestine is responsible for the digestion of carbohydrates. Inhibiting this enzyme by preventing the digestion of carbohydrates can reduce postprandial hyperglycemia. Designing and reporting new compounds with coumarin and triazole core, using virtual screening, is an optimal, simple and low-cost method and can be useful in inhibiting alpha-glucosidase enzyme. Materials and Methods: First, the crystallographic structure of alpha-glucosidase enzyme with identifier code 3A4A was obtained from Protein Data Bank (PDB). Then, a library of 1922 chemical compounds that were structurally similar to the main triazole and coumarin skeletons was formed from the PubChem database. Various filters were applied to compounds by PyRx0.8 software and Molinspiration, SwissADME and admetSAR servers. Finally, the binding energy and interaction of the compounds were checked by Autodock 4.2 software. Results: Among the compounds in the library, eight compounds passed the mentioned filters successfully. Then, molecular docking was performed on these compounds. Among the eight docked compounds, four compounds with the identification codes of CID_132277006, CID_1534998, CID_110745226 and CID_122233777 were introduced as the best compounds with binding energy of -9.19, -8.89, -8.71 and -8.49 kcal/mol respectively. Examining the results of molecular docking showed that amino acids Arg442, Gln279, Ser241, His351, Lys156, Asp352, Glu277, Arg315, Asp242, Phe303, Ser157, Asn415, Glu411, Phe314, Tyr158, Ser240, Asp215 and Val216 have a higher importance in the formation of compounds complex with the active site of alpha-glucosidase enzyme. Conclusion: It seems that the hydrophobic bond and on the next level the hydrogen bond, then the cation-π bond are important for the interaction of the compounds with the active site of alpha-glucosidase enzyme.
| نوع سند : | پایان نامه (دکتری حرفه ای ) |
|---|---|
| زبان سند : | فارسی |
| استاد راهنما : | ساقی سپهری |
| استاد مشاور : | زهره باخرد |
| دانشجو : | فرشته استانی |
| کلیدواژه ها (فارسی): | آلفا-گلوکوزیداز، کومارین، تریازول، غربالگری مجازی، داکینگ مولکولی |
| کلیدواژه ها (انگلیسی): | alpha-glucosidase, coumarin, triazole, virtual screening, molecular docking |
| موضوعات : | QV فارماکولوژی |
| بخش های دانشگاهی : | دانشکده داروسازی > بخش شیمی دارویی دانشکده داروسازی > واحد پژوهش - پایان نامه های دفاع شده |
| کد شناسایی : | 16756 |
| ارائه شده توسط : | آقای فرهاد خدایی |
| ارائه شده در تاریخ : | 18 بهمن 1401 13:45 |
| آخرین تغییر : | 18 بهمن 1401 13:45 |
فقط پرسنل کتابخانه صفحه کنترل اسناد